Muitos tipos diferentes de bactérias e vírus podem causar pneumonia, mas não há uma maneira fácil de determinar qual o micróbio que está causando a doença em um determinado paciente. Essa incerteza torna mais difícil para os médicos escolherem os tratamentos eficazes, porque os antibióticos comumente usados para tratar a pneumonia bacteriana não ajudam os pacientes que estejam com pneumonia viral.
Além disso, não utilizar antibióticos indiscriminadamente é um passo importante para reduzir a resistência das bactérias a esses medicamentos.
Para tentar solucionar esse problema, pesquisadores do MIT estão projetando um sensor que poderia distinguir entre as infecções por pneumonia viral ou bacteriana. Através deste novo método de diagnóstico, os pesquisadores esperam colaborar para evitar que os médicos prescrevam antibióticos nos casos em que eles não serão eficazes.
“O desafio é que existem muitos patógenos diferentes que podem levar a diferentes tipos de pneumonia e, mesmo com os testes mais extensos e avançados, o patógeno específico que causa a doença de alguém não pode ser identificado em cerca de metade dos pacientes. Você trata uma pneumonia viral com antibióticos e então você pode estar contribuindo para a resistência à antibióticos, o que é um grande problema e o paciente não vai melhorar”, diz Sangeeta Bhatia, professor de Ciências e Tecnologia da Saúde de John e Dorothy Wilson, de Engenharia Elétrica e Ciência da Computação no MIT e membro do Koch Institute for Integrative Cancer Research.
Uma razão pela qual tem sido difícil distinguir entre a pneumonia viral e bacteriana é que existem muitos micróbios que podem causar pneumonia, incluindo as bactérias Streptococcus pneumoniae e Haemophilus influenzae , e vírus como influenza e vírus sincicial respiratório (RSV).
Em um estudo com camundongos, usando um simples teste de urina para ler os resultados, os pesquisadores mostraram que os sensores podiam distinguir com precisão a pneumonia bacteriana da viral em cerca de duas horas,
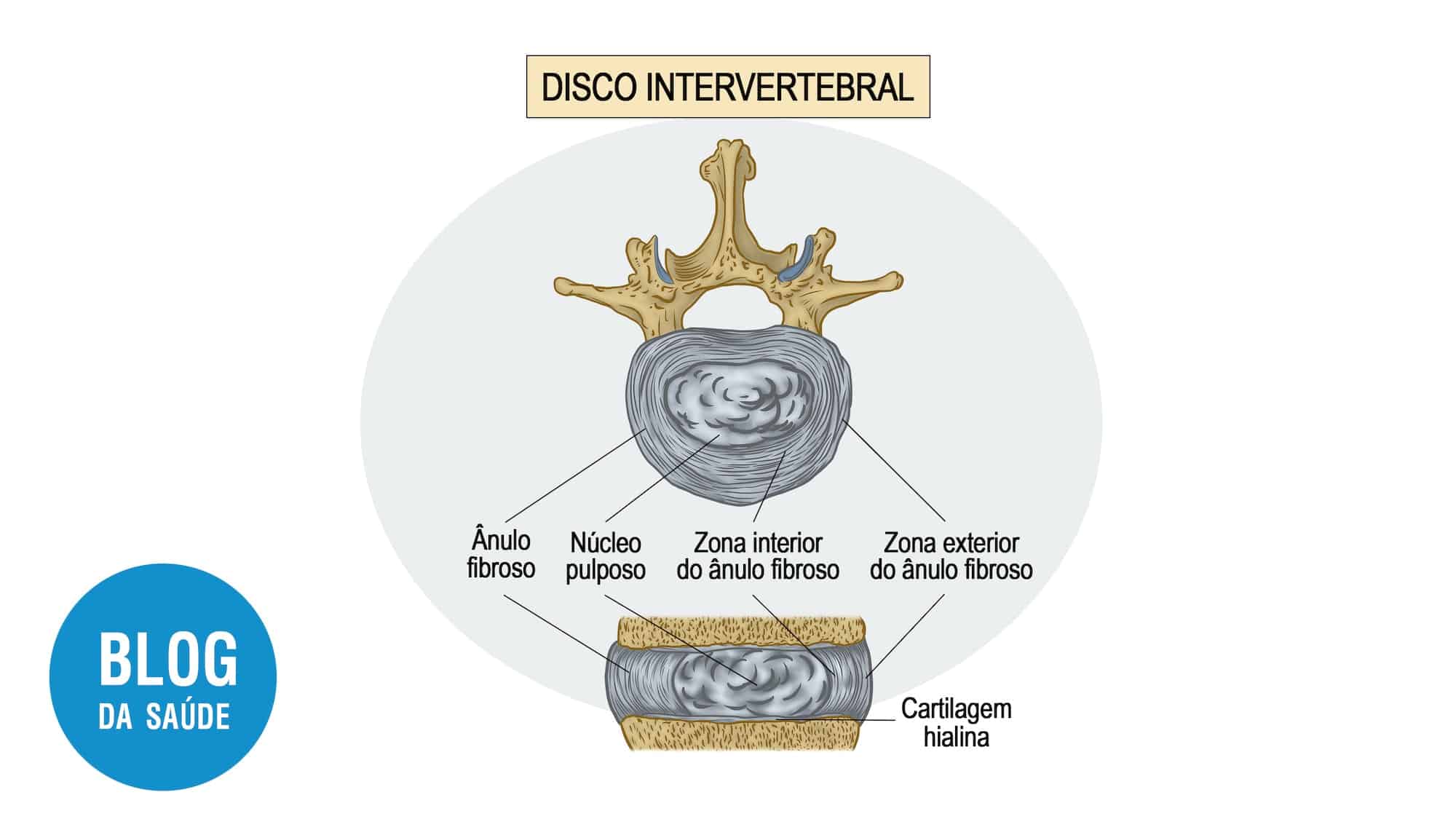
Ao projetar seu sensor, a equipe de pesquisa decidiu se concentrar em medir a resposta do hospedeiro à infecção, em vez de tentar detectar o próprio patógeno. Infecções virais e bacterianas provocam tipos distintos de respostas imunes, que incluem a ativação de enzimas chamadas proteases, que quebram proteínas. A equipe do MIT descobriu que o padrão de atividade dessas enzimas pode servir como uma assinatura de infecção bacteriana ou viral.
O genoma humano codifica mais de 500 proteases, e muitas delas são usadas por células que respondem à infecção, incluindo células T, neutrófilos e células natural killer (NK). Uma equipe liderada por Purvesh Khatri, professor associado de medicina e ciência de dados biomédicos da Universidade de Stanford e um dos autores do artigo, coletou 33 conjuntos de dados disponíveis publicamente de genes que são expressos durante infecções respiratórias. Ao analisar esses dados, Khatri conseguiu identificar 39 proteases que parecem responder de maneira diferente a diferentes tipos de infecção.
Bhatia e seus alunos usaram esses dados para criar 20 sensores diferentes que podem interagir com essas proteases. Os sensores consistem em nanopartículas revestidas com peptídeos que podem ser clivados por proteases particulares. Cada peptídeo é marcado com uma molécula portadora que é liberada quando os peptídeos são clivados por proteases que são reguladas positivamente na infecção. Esses portadores são eventualmente excretados na urina. A urina pode então ser analisada com espectrometria de massa para determinar quais proteases são mais ativas nos pulmões.
Os pesquisadores testaram seus sensores em cinco modelos de camundongos diferentes de pneumonia, causada por infecções de Streptococcus pneumoniae , Klebsiella pneumoniae , Haemophilus influenzae , vírus da gripe e vírus da pneumonia de camundongos.
Depois de ler os resultados dos testes de urina, os pesquisadores usaram aprendizado de máquina para analisar os dados. Usando essa abordagem, eles foram capazes de treinar algoritmos que poderiam diferenciar entre pneumonia e controles saudáveis, e também distinguir se uma infecção era viral ou bacteriana, com base nesses 20 sensores.
Os pesquisadores também descobriram que seus sensores podiam distinguir entre os cinco patógenos testados, mas com menor precisão do que o teste para distinguir entre vírus e bactérias. Uma possibilidade que os pesquisadores podem buscar é desenvolver algoritmos que possam não apenas distinguir infecções bacterianas de infecções virais, mas também identificar a classe de micróbios que causam uma infecção bacteriana, o que pode ajudar os médicos a escolher o melhor antibiótico para combater esse tipo de bactéria.
A leitura baseada na urina também é passível de detecção futura com uma tira de papel, semelhante a um teste de gravidez, o que permitiria o diagnóstico no local de atendimento. Para esse fim, os pesquisadores identificaram um subconjunto de cinco sensores que poderiam colocar os testes em casa mais próximos. No entanto, é necessário mais trabalho para determinar se o painel reduzido funcionaria da mesma forma em humanos, que possuem mais variabilidade genética e clínica do que os camundongos.
Padrões de resposta
No estudo, os pesquisadores também identificaram alguns padrões de resposta do hospedeiro a diferentes tipos de infecção. Em camundongos com infecções bacterianas, as proteases secretadas pelos neutrófilos foram observados com mais destaque, o que era esperado porque os neutrófilos tendem a responder mais às infecções bacterianas do que às infecções virais.
As infecções virais, por outro lado, provocaram atividade de protease das células T e células NK, que geralmente respondem mais às infecções virais. Um dos sensores que gerou o sinal mais forte estava ligado a uma protease chamada granzima B, que desencadeia a morte celular programada. Os pesquisadores descobriram que esse sensor foi altamente ativado nos pulmões de camundongos com infecções virais e que tanto as células NK quanto as T estavam envolvidas na resposta.
Para entregar os sensores em camundongos, os pesquisadores os injetaram diretamente na traqueia, mas agora estão desenvolvendo versões para uso humano que podem ser administradas usando um nebulizador ou um inalador semelhante ao inalador de asma. Eles também estão trabalhando para encontrar uma maneira de detectar os resultados usando um bafômetro em vez do teste de urina, o que pode oferecer resultados ainda mais rápidos.
A pesquisa foi financiada, em parte, pela Fundação Bill e Melinda Gates, Janssen Research and Development, Koch Institute Support (core) Grant do National Cancer Institute e do National Institute of Environmental Health Sciences.
Confira o Estudo : 10.1073/pnas.2121778119